LabChip GX Touch 核酸解析装置



Pick a product variant
LabChip GX Touch nucleic acid analyzer benefits
Ease of use
High throughput
Flexible
Small footprint
Cost efficient
Fast analysis
Increased efficiency
Supports testing in a streamlined and reproducible GMP-compliant environment
Coming soon... Empower on LabChip!
Take full control of your nucleic acid and protein assays on the LabChip GX Touch and LabChip GXII Touch systems with Waters® Empower™ software. Seamlessly acquire and analyze your data with a single software with the new LabChip Empower™ v1.0 driver.
Improved efficiency
By running LabChip systems directly from the Empower software, labs can streamline workflows, reduce hands-on time, and boost productivity.
Data integrity
Ensure your data is securely stored and readily available for review, analysis, and audit through the Empower software’s robust compliance features.
Future-proof
LabChip Empower driver v1.0 is fully compatible with the Empower software, ensuring your system is ready for future software updates and advancements.
Video overview
DNA and RNA assays to address your needs
- Plasmid DNA assay
- DNA 1K assay
- DNA 5K assay
- DNA 12K assay
- DNA high sensitivity assay
- Genomic DNA assay
- NGS 3K assay
- cfDNA assay
- RNA standard RNA assay
- Pico RNA assay
- Small RNA assay
- DNA high sensitivity assay
Product information
Overview
DNA, RNA, NGSライブラリーの正確なサイズ測定, 定量化, , 品質管理
LabChip® GX Touch™ 核酸解析装置は、ゲノムのサイズ測定と定量化に対応する包括的なソリューションです。以下の利点を提供します:
- DNAおよびRNAサンプルの完全性の定量をアガロースゲルよりも数時間速く実現
- cfDNA、PCRフリーライブラリー、ChIP-seqライブラリーなどの希少または低濃度サンプルを最大限に活用
- DNAスメアでは25 pg/µL、フラグメントでは0.5 pg/µLまでの正確な定量化を実現
- NGSライブラリーの調製解析および品質管理
- 96ウェルまたは384ウェル(HT)フォーマットにおける費用効率の良い柔軟なスループットオプション
- エレクトロフェログラム、バーチャルゲル、表形式レポートなどのデータ視覚化オプション
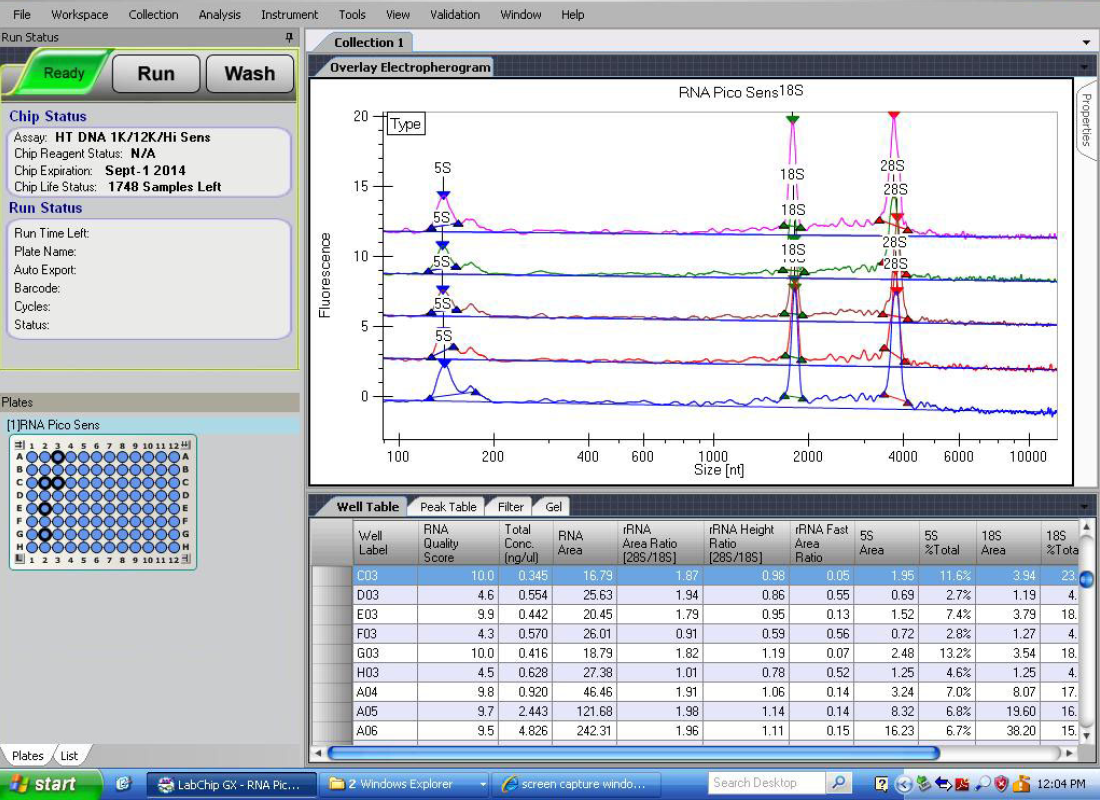
LabChip GX Touch核酸アナライザーは、さまざまなDNAおよびRNA解析にも対応しており、最小限の入力要件で比類のないゲノムデータ品質を提供します。濃度が低く、サンプルが貴重な場合、LabChip® DNA High Sens および RNA Pico アッセイは、信頼性の高い測定とサンプル保存に最適な選択肢です。約30秒でゲノム材料の完全な解析を行うLabChip GX Touch核酸分析装置は、核酸定量ワークフローのボトルネックを解消します。高解像度解析により、わずか2 pg/µLのサンプルから5 bpまでのDNAフラグメントやスメアの正確なサイズ測定と定量化が可能です。
システムは幅広いDNA解析オプションを提供しており、中でもNGS 3Kアッセイは、最小限のサンプル投入量で比類のないゲノムデータ品質を提供します。この高い感度は、濃度が低く、サンプルが貴重で、それらの保存が不可欠である場合に、gDNAやPCR非依存DNA定量の理想的なツールとなります。
Coming soon ….Empower on LabChip!
Waters Empower™ ソフトウェアを使用することで、LabChip GX Touch™ および LabChip GXII Touch™ システム上で核酸およびタンパク質アッセイを自在に管理できます。新しいLabChip™ Empower™ v1.0ドライバーにより、ひとつのソフトウェアでデータの取得から解析までをシームレスに実行できます。
”request more information” ボタンから営業担当者にお問い合わせいただくと、LabChip Empowerドライバー v1.0へのアップグレード方法や、以下の機能についてご案内いたします:
- 効率性の向上: EmpowerソフトウェアでLabChipシステムを直接運用することで、ワークフローを合理化し、作業時間を削減し、生産性を高めることができます。
- データの完全性: Empowerソフトウェアの堅牢なコンプライアンス機能を通じて、データを安全に保存し、レビュー・解析・監査にすぐに活用できます。
- 将来性: LabChip Empower ドライバー v1.0 は Empowerソフトウェアと完全に互換性があり、今後のソフトウェアアップデートや進展に対応できるシステムを実現します。
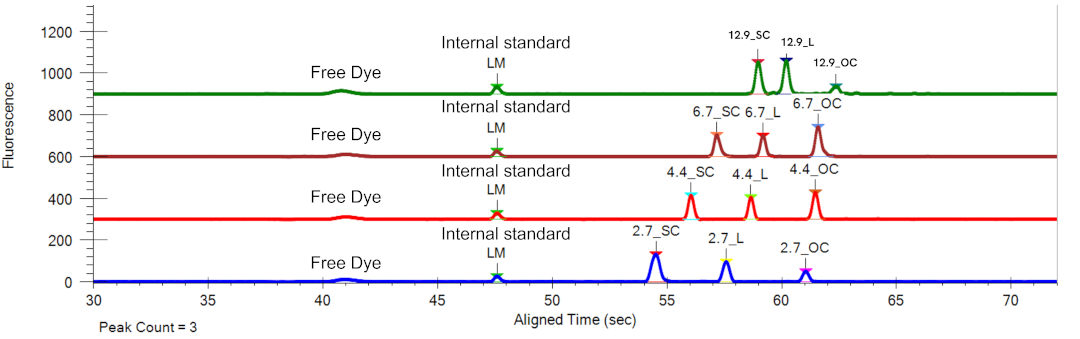
mRNAベースワクチンの純度と完全性解析
LabChip® RNA Assay を使用することで、メルクの研究者たちはmRNAワクチンの純度と完全性を特徴付けるアッセイを開発しました。この迅速で簡単かつ効率的なワークフローは、ワクチン開発のスピードを上げ、増加する大量製造の需要に応えるのに役立ちました。
システムは、多様なDNA解析オプションを提供しており、NGS 3KアッセイやcfDNAアッセイを含みます。これらは、最小限の入力要件で比類のないゲノムデータ品質を実現します。この高い感度により、濃度が低く、サンプルが貴重で保存性が重要となる場合のgDNA、cfDNA、およびPCRフリーDNAの定量に最適なツールとなります。
外形寸法
- 高さ 69 cm
- 幅 51 cm
- 奥行き 49 cm
Specifications
| Dimensions | 51.0 cm (W) x 49.0 cm (D) |
|---|
| 21CFR Compatible |
Yes
|
|---|---|
| Brand |
LabChip GX
|
| Model |
LabChip GX Touch 24 核酸解析装置
|
| Sample Quantity |
48
|
| Throughput |
ロースループット
|
- Bagheri, H., Friedman, H., Shao, H., Chong, Y., Lo, C., Emran, F., . . . Peterson, A. (2018). TIE: A Method to Electroporate Long DNA Templates into Preimplantation Embryos for CRISPR-Cas9 Gene Editing. The CRISPR Journal,1(3), 223-229. doi:10.1089/crispr.2017.0020.
- Chesnais, V., Ott, A., Chaplais, E., Gabillard, S., Pallares, D., Vauloup-Fellous, C., . . . Ginoux, E. (2018). Using massively parallel shotgun sequencing of maternal plasmatic cell-free DNA for cytomegalovirus DNA detection during pregnancy: A proof of concept study. Scientific Reports, 8(1). doi:10.1038/s41598-018-22414-6.
- Costa, J-M., et al. (2018). Cell-free fetal DNA versus maternal serum screening for trisomy 21 in pregnant women with and without assisted reproduction technology: a prospective interventional study. Genetics in Medicine. doi:10.1038/gim.2018.4.
- Faiz, A., Heijink, I. H., Vermeulen, C. J., Guryev, V., Berge, M. V., Nawijn, M. C., & Pouwels, S. D. (2018). Cigarette smoke exposure decreases CFLAR expression in the bronchial epithelium, augmenting susceptibility for lung epithelial cell death and DAMP release. Scientific Reports,8(1). doi:10.1038/s41598-018-30602-7.
- Ford, L., Carter, G. P., Wang, Q., Seemann, T., Sintchenko, V., Glass, K., . . . Kirk, M. D. (2018). Incorporating Whole-Genome Sequencing into Public Health Surveillance: Lessons from Prospective Sequencing of Salmonella Typhimurium in Australia. Foodborne Pathogens and Disease, 15(3), 161-167. doi:10.1089/fpd.2017.2352.
- Gergen, J., Coulon, F., Creneguy, A., Elain-Duret, N., Gutierrez, A., Pinkenburg, O., . . . Haspot, F. (2018). Multiplex CRISPR/Cas9 system impairs HCMV replication by excising an essential viral gene. Plos One, 13(2). doi:10.1371/journal.pone.0192602.
- Giand et al. (2021) Early transmission of SARS-CoV-2 in South Africa: An epidemiological and phylogenetic report. International Journal of Infectious Diseases, 103, 234-241, doi.org/10.1016/j.ijid.2020.11.128.
- Kim, K.W., et al. (2021) Respiratory viral co-infections among SARS-CoV-2 cases confirmed by virome capture sequencing. Sci Rep 11, 3934 doi.org/10.1038/s41598-021-83642-x.
- Mogilevsky, M., Shimshon, O., Kumar, S., Mogilevsky, A., Keshet, E., Yavin, E., Heyd, F., and Karni, R. (2018) Modulation of MKNK2 alternative splicing by splice-switching oligonucleotides as a novel approach for glioblastoma treatment. Nucleic Acids Res. doi: 10.1093/nar/gky921.
- Ong, J., Woldhuis, R. R., Boudewijn, I. M., Berg, A. V., Kluiver, J., Kok, K., . . . Brandsma, C. A. (2019). Age-related gene and miRNA expression changes in airways of healthy individuals. Scientific Reports,9(1). doi:10.1038/s41598-019-39873-0.
- Raffaele, J., Loughney, J. W., & Rustandi, R. R. (2021). Development of a microchip capillary electrophoresis method for determination of the purity and integrity of mrna in lipid nanoparticle vaccines. ELECTROPHORESIS. doi.org/10.1002/elps.202100272.
- Trolle, C., et al. (2016) Widespread DNA hypomethylation and differential gene expression in Turner syndrome. Scientific Reports (6). doi.org/10.1038/srep34220.
Citations
Resources
Are you looking for resources, click on the resource type to explore further.
Assess RNA quality and integrity with the LabChip GX Touch system for precise electrophoretic separation, real-time analysis, and...
The need for better profiling is clear, and technological advances are making it possible to optimize clinical trials and...
Learn how the LabChip systems automate DNA, RNA, & protein analysis with GxP Software for compliance, access control, & data...


How can we help you?
We are here to answer your questions.